TGuide Blutgenomisches DNA-Kit
Anwendungen
Das gereinigte Genom kann direkt in PCR, RT-PCR, Enzymverdauungsreaktion, Southern-Hybridisierung und anderen Experimenten verwendet werden.
Merkmale
■ Einfache und schnelle Extraktion: TGuide-Zubehörprodukte basieren auf dem Prinzip der Nukleinsäurereinigung durch magnetische Beads und die Extraktion der genomischen DNA kann in 44 min abgeschlossen werden.
■ Zuverlässige Ergebnisse: Die mit dem Kit extrahierte genomische DNA enthält keine Verunreinigungen wie RNA und Protein und kann direkt für die PCR oder quantitative Fluoreszenz-PCR verwendet werden.
■ Keine Phenol- und Chloroform-Extraktion: Es werden keine für den menschlichen Körper schädlichen organischen Lösungsmittel wie Phenol und Chloroform benötigt.
■ Flexible Anfangsprobengröße: 200 μl, 400 μl, 1,2 ml Vollblut oder 1-3 ml vorseparierte Leukozyten können durch Auswahl des entsprechenden Kits direkt extrahiert werden.
Alle Produkte können für ODM/OEM angepasst werden. Für Details,Bitte klicken Sie auf kundenspezifischen Service (ODM/OEM)
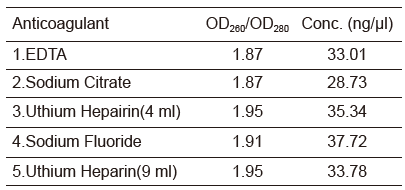 |
Ergebnisse genomischer DNA, extrahiert aus 200 μl Vollblut, das mit verschiedenen Antikoagulanzien behandelt wurde |
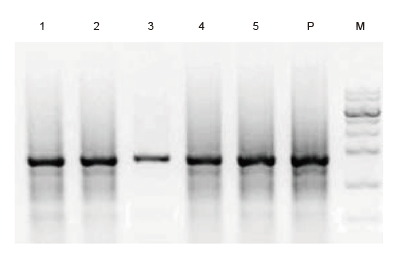 |
DNA wurde in 200 µl Elutionspuffer gelöst. M: 1-kb-Leiter 1-5 Nachweis von Inhibitoren verschiedener Antikoagulanzien. Zielgen: Cbl-b-Gen, 1,7 kb P: Positivkontrolle |
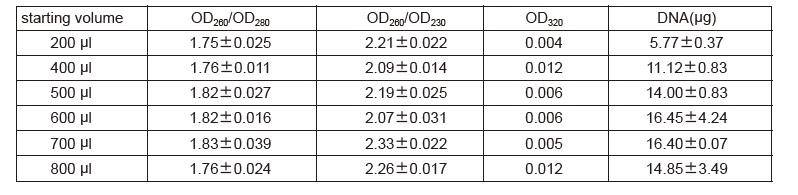
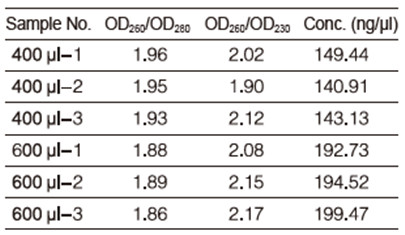 |
Ergebnisse genomischer DNA, extrahiert aus 400 μl oder 600 μl Vollblut |
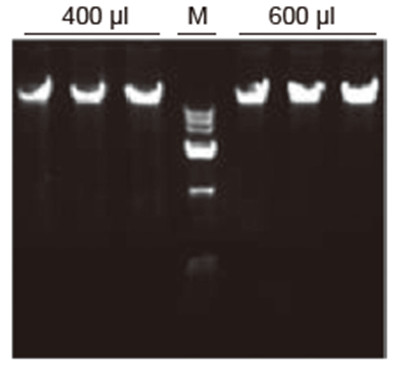 |
DNA wurde in 100 µl Elutionspuffer gelöst Genomische DNA extrahiert aus 400 μl oder 600 μl Vollblut M: DL15000 Marker Ladevolumen: 2 μl |
 |
Ergebnisse der aus 1,2 ml Vollblut extrahierten genomischen DNA |
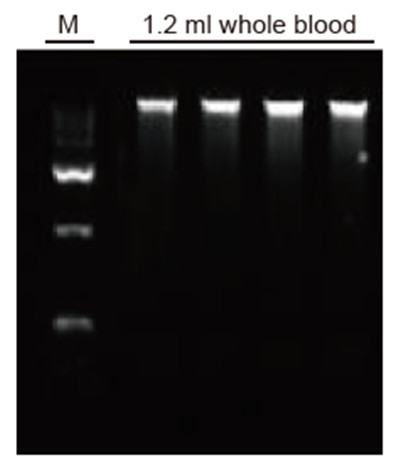 |
DNA wurde in 300 µl Elutionspuffer gelöst Genomische DNA extrahiert aus 1,2 ml Vollblut M: DL15000-Marker; Ladevolumen: 2 μl |
A-1 Niedrige Konzentration von Zellen oder Viren in der Ausgangsprobe – Erhöhen Sie die Konzentration von Zellen oder Viren.
A-2 Unzureichende Lyse der Proben —Die Proben wurden nicht gründlich mit dem Lysepuffer vermischt. Es wird empfohlen, 1-2 Mal durch Pulsvortexen gründlich zu mischen. —Unzureichende Zelllyse durch die Aktivitätsabnahme der Proteinase K. —Unzureichende Zelllyse oder Proteinabbau aufgrund unzureichender Warmbadzeit. Es wird empfohlen, das Gewebe in kleine Stücke zu schneiden und die Badezeit zu verlängern, um alle Rückstände im Lysat zu entfernen.
A-3 Unzureichende DNA-Adsorption. – Es wurde kein Ethanol oder ein niedriger Prozentsatz anstelle von 100 % Ethanol zugegeben, bevor das Lysat auf die Spin-Säule übertragen wurde.
A-4 Der pH-Wert des Elutionspuffers ist zu niedrig. —Stellen Sie den pH-Wert zwischen 8,0 und 8,3 ein.
Restethanol im Eluenten.
— Im Eluenten befindet sich noch Waschpuffer PW. Das Ethanol kann durch Zentrifugieren der Spinsäule für 3–5 Minuten und anschließendes Platzieren bei Raumtemperatur oder einem 50 °C-Inkubator für 1-2 Minuten entfernt werden.
A-1 Die Probe ist nicht frisch. – Extrahieren Sie eine positive Proben-DNA als Kontrolle, um festzustellen, ob die DNA in der Probe abgebaut wurde.
A-2 Unsachgemäße Vorbehandlung. —Verursacht durch übermäßiges Mahlen mit flüssigem Stickstoff, Feuchtigkeitsrückgewinnung oder zu große Probenmenge.
Die Vorbehandlungen sollten für verschiedene Proben variieren. Stellen Sie bei Pflanzenproben sicher, dass sie in flüssigem Stickstoff gründlich gemahlen werden. Bei Tierproben homogenisieren oder gründlich in flüssigem Stickstoff mahlen. Bei Proben mit schwer aufzubrechenden Zellwänden, wie G+ Bakterien und Hefen, wird empfohlen, Lysozym, Lyticase oder mechanische Methoden zum Aufbrechen der Zellwände zu verwenden.
4992201/4992202 Plant Genomic DNA Kit verwendet eine säulenbasierte Methode, die Chloroform für die Extraktion erfordert. Es eignet sich besonders für verschiedene Pflanzenproben sowie Pflanzentrockenpulver. Das Hi-DNAsecure Plant Kit ist ebenfalls säulenbasiert, erfordert jedoch keine Phenol-/Chloroform-Extraktion, was es sicher und ungiftig macht. Es ist für Pflanzen mit hohem Polysaccharid- und Polyphenolgehalt geeignet. 4992709/4992710 DNAquick Plant System verwendet eine flüssigkeitsbasierte Methode. Eine Phenol/Chloroform-Extraktion ist ebenfalls nicht erforderlich. Das Reinigungsverfahren ist einfach und schnell ohne Begrenzung der Probenstartmengen, sodass Benutzer die Menge flexibel an die experimentellen Anforderungen anpassen können. Große gDNA-Fragmente können mit hoher Ausbeute erhalten werden.
Die Blutgerinnsel-DNA-Extraktion kann unter Verwendung der in diesen beiden Kits enthaltenen Reagenzien durchgeführt werden, indem einfach das Protokoll auf die spezifischen Anweisungen für die Blutgerinnsel-DNA-Extraktion geändert wird. Die Softcopy des DNA-Extraktionsprotokolls für Blutgerinnsel kann auf Anfrage ausgestellt werden.
Suspendieren Sie die frische Probe mit 1 ml PBS, normaler Kochsalzlösung oder TE-Puffer. Die Probe mit einem Homogenisator vollständig homogenisieren und den Niederschlag durch Zentrifugieren am Boden eines Röhrchens sammeln. Entsorgen Sie den Überstand und resuspendieren Sie das Präzipitat mit 200 ul Puffer GA. Die folgende DNA-Reinigung kann gemäß der Anleitung durchgeführt werden.
Für die Aufreinigung von gDNA in Plasma-, Serum- und Körperflüssigkeitsproben wird das TIANamp Micro DNA Kit empfohlen. Für die Aufreinigung von Virus-gDNA aus Serum-/Plasmaproben wird das TIANamp Virus DNA/RNA Kit empfohlen. Für die Reinigung bakterieller gDNA aus Serum- und Plasmaproben wird das TIANamp Bacteria DNA Kit empfohlen (bei positiven Bakterien sollte Lysozym enthalten sein). Für Speichelproben werden das Hi-Swab DNA Kit und das TIANamp Bacteria DNA Kit empfohlen.
DNAsecure Plant Kit oder DNAquick Plant System werden für die Extraktion des Pilzgenoms empfohlen. Für die Hefegenomextraktion wird das TIANamp Yeast DNA Kit empfohlen (Lyticase sollte selbst hergestellt werden).
Produktkategorien
WARUM UNS WÄHLEN
Seit seiner Gründung entwickelt unsere Fabrik erstklassige Produkte unter Einhaltung des Prinzips
der Qualität zuerst. Unsere Produkte haben einen ausgezeichneten Ruf in der Branche und ein wertvolles Vertrauen bei neuen und alten Kunden gewonnen.