RNAprep Pure Cell/Bakterien-Kit
Merkmale
■ Optimierte Puffer und Protokolle für kultivierte Zellen und Bakterienproben machen den Prozess einfach und bequem.
■ Einzigartige DNase I minimiert die Kontamination mit genomischer DNA.
■ Einzigartige RNase-freie Filtrationssäulen CS beseitigen andere Kontaminationen.
■ Die hochreine und gebrauchsfertige RNA eignet sich für sensible Downstream-Anwendungen.
■ Keine Phenol-/Chloroform-Extraktion, keine LiCl- und Ethanol-Fällung, keine CsCl-Gradientenzentrifugation erforderlich, was den Prozess sicher und zuverlässig macht.
Anwendungen
■ RT-PCR.
■ Northern-Blot, Dot-Blot.
■ Echtzeit-PCR.
■ Chipanalyse.
■ PolyA-Screening, In-vitro-Translation, RNase-Schutzanalyse und molekulares Klonen.
Alle Produkte können für ODM/OEM angepasst werden. Für Details,Bitte klicken Sie auf kundenspezifischen Service (ODM/OEM)
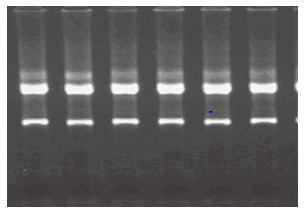 |
Material: Menschliche Jurkat-Zellen (1×10 .)6 ) Methode: Die Gesamt-RNA von humanen Jukat-Zellen wurde mit dem RNAprep Pure Cell/Bacteria Kit isoliert. Ergebnisse: Siehe obiges Bild der Agarosegelelektrophorese. Pro Spur wurden 2-4 µl 50 µl Eluate geladen. Die Elektrophorese wurde 30 min bei 6 V/cm auf 1% Agarosegel durchgeführt. |
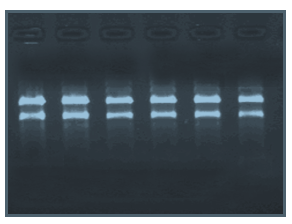 |
Material: TOP10 E.coli (1×10 .)8) Methode: Die Gesamt-RNA von TOP10 E.coli wurde mit dem RNAprep Pure Cell/Bacteria Kit isoliert. Ergebnisse: Siehe obiges Bild der Agarosegelelektrophorese. Pro Spur wurden 2-4 µl 50 µl Eluate geladen. Die Elektrophorese wurde 30 min bei 6 V/cm auf 1% Agarosegel durchgeführt. |
A-1 Zelllyse oder Homogenisierung nicht ausreichend
---- Reduzieren Sie den Probenverbrauch, erhöhen Sie die Menge an Lysepuffer, erhöhen Sie die Homogenisierungs- und Lysezeit.
A-2 Probenmenge ist zu groß
---- Verringern Sie die verwendete Probenmenge oder erhöhen Sie die Menge an Lysepuffer.
A-1 Unzureichende Zelllyse oder Homogenisierung
---- Reduzieren Sie den Probenverbrauch, erhöhen Sie die Menge an Lysepuffer, erhöhen Sie die Homogenisierungs- und Lysezeit.
A-2 Probenmenge ist zu groß
----Bitte beachten Sie die maximale Verarbeitungskapazität.
A-3 RNA wird nicht vollständig von der Säule eluiert
---- Nach Zugabe von RNase-freiem Wasser einige Minuten ruhen lassen, bevor zentrifugiert wird.
A-4 Ethanol im Eluenten
---- Nach dem Spülen erneut zentrifugieren und Waschpuffer so weit wie möglich entfernen.
A-5 Zellkulturmedium wird nicht vollständig entfernt
---- Achten Sie beim Sammeln von Zellen darauf, das Kulturmedium so weit wie möglich zu entfernen.
A-6 Die in RNAstore gelagerten Zellen werden nicht effektiv zentrifugiert
---- Die RNAstore-Dichte ist höher als das durchschnittliche Zellkulturmedium; Daher sollte die Zentrifugalkraft erhöht werden. Es wird empfohlen, bei 3000x g zu zentrifugieren.
A-7 Niedriger RNA-Gehalt und -Häufigkeit in der Probe
---- Verwenden Sie eine positive Probe, um festzustellen, ob die geringe Ausbeute durch die Probe verursacht wird.
A-1 Das Material ist nicht frisch
---- Frisches Gewebe sollte sofort in flüssigem Stickstoff aufbewahrt oder sofort in das RNAstore-Reagenz gegeben werden, um den Extraktionseffekt sicherzustellen.
A-2 Probenmenge ist zu groß
---- Probenmenge reduzieren.
A-3 RNase-Kontaminationn
----Obwohl der im Kit enthaltene Puffer keine RNase enthält, kann RNase während des Extraktionsprozesses leicht kontaminiert werden und sollte mit Vorsicht behandelt werden.
A-4 Elektrophorese-Verschmutzung
---- Ersetzen Sie den Elektrophoresepuffer und stellen Sie sicher, dass die Verbrauchsmaterialien und der Ladepuffer frei von RNase-Kontaminationen sind.
A-5 Zu hohe Belastung für Elektrophorese
---- Reduzieren Sie die Menge der Probenbeladung, die Beladung jedes Wells sollte 2 μg nicht überschreiten.
A-1 Probenmenge ist zu groß
---- Probenmenge reduzieren.
A-2 Einige Proben haben einen hohen DNA-Gehalt und können mit DNase behandelt werden.
----Führen Sie eine RNase-freie DNase-Behandlung mit der erhaltenen RNA-Lösung durch, und die RNA kann nach der Behandlung direkt für nachfolgende Experimente verwendet oder durch RNA-Reinigungskits weiter gereinigt werden.
Für Glaswaren, 4 h bei 150°C gebacken. Bei Kunststoffbehältern 10 min in 0,5 M NaOH eingetaucht, dann gründlich mit RNase-freiem Wasser gespült und dann sterilisiert, um RNase vollständig zu entfernen. Die im Experiment verwendeten Reagenzien oder Lösungen, insbesondere Wasser, müssen frei von RNase sein. Verwenden Sie RNase-freies Wasser für alle Reagenzienzubereitungen (Wasser in eine saubere Glasflasche geben, DEPC bis zu einer Endkonzentration von 0,1% (V/V) hinzufügen, über Nacht schütteln und autoklavieren).
Produktkategorien
WARUM UNS WÄHLEN
Seit seiner Gründung entwickelt unsere Fabrik erstklassige Produkte unter Einhaltung des Prinzips
der Qualität zuerst. Unsere Produkte haben einen ausgezeichneten Ruf in der Branche und ein wertvolles Vertrauen bei neuen und alten Kunden gewonnen.