FastKing RT-Kit (mit gDNase)
Merkmale
■ Hohe Effizienz: Das FastKing RT-Enzym ist mit einem hydrophoben Motiv modifiziert, mit einer RT-Effizienz von mehr als 95 %.
■ Sensibel: Vorlagen mit nur 1 ng können genau identifiziert werden.
■ Resistenz: Reverse Transkription komplexer Template mit perfekter Resistenz gegen Verunreinigungen.
■ Flexibel: Die Entfernung der genomischen DNA und die reverse Transkription wurden separat durchgeführt. Primer wurden separat in einem Röhrchen gemischt, um flexibel andere Primer zu wechseln.
Spezifikation
Typ: Genmodifizierte Reverse Transkriptase, gDNase
Verfahren: Zweistufig (genomische DNA-Entfernung und RT)
RT-Effizienz: >95%
Vorlage: 1 ng - 2 μg
Betriebszeit: ~21 min
Anwendungen: Die revers transkribierte cDNA kann in konventioneller PCR, Real time PCR, cDNA-Bibliotheksaufbau verwendet werden.
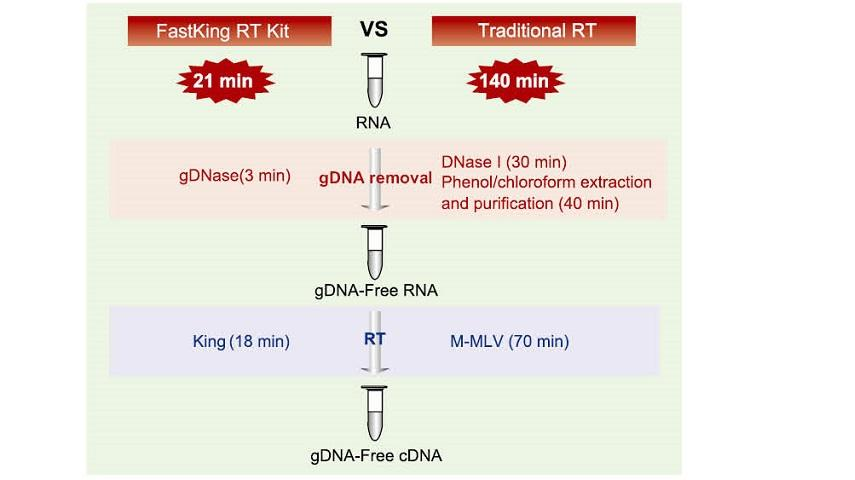
21 min Reaktion in einem Röhrchen
Es dauert nur 21 Minuten, um die gDNA-Entfernung und den effizienten reversen Transkriptionsprozess im selben Röhrchen abzuschließen, ohne das Reaktionsröhrchen und den unabhängigen DNase-I-Behandlungsprozess zu ersetzen. Verglichen mit der traditionellen Methode, die einen 12-Schritt-Betrieb und eine 140-minütige Reaktion erfordert, vereinfacht sie die Arbeitsschritte erheblich und spart viel Betriebszeit.
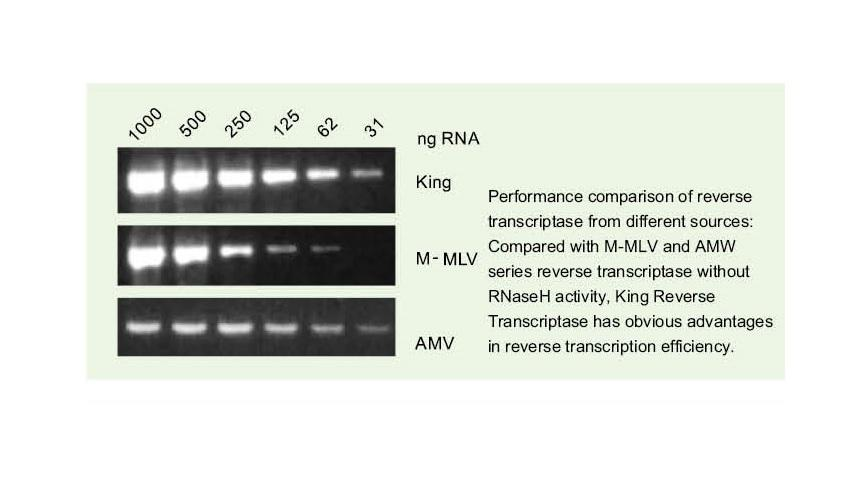
Hervorragende Qualität von King RTase
——Ultrahohe Effizienz der reversen Transkription
——Die Effizienz der reversen Transkription beträgt über 95 %
Die allgemeine reverse Transkriptase hat eine reverse Transkriptionseffizienz von 40-60% und die cDNA-Ausbeute kann durch eine höhere RNA-Beladungsmenge erhöht werden. King Reverse Transkriptase kann aufgrund ihrer einzigartigen hohen Affinität für RNA-Matrizen eine reverse Transkriptionseffizienz von mehr als 95 % erreichen. Daher können nachfolgende Experimente ohne die Notwendigkeit einer großen Menge an RNA-Eingabe durchgeführt werden, was RNA einspart und eine hohe Reinheit und eine hohe Ausbeute an cDNA ermöglicht.
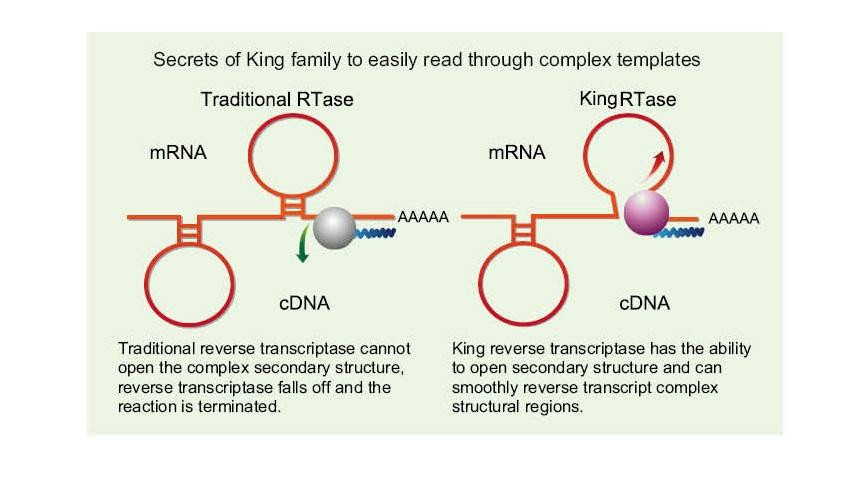
Einfaches Lesen komplexer Vorlagen
——Leicht durch hohe GC und komplexe Vorlagen zu lesen
Einzelsträngige RNA weist aufgrund von Wasserstoffbrückenbindungen zwischen den Strängen eine breite Palette komplexer Sekundärstrukturregionen auf. Gewöhnliche reverse Transkriptase kann zu einer Beendigung der reversen Transkription führen, wenn sie auf eine komplexe Sekundärstruktur trifft, wodurch die cDNA-Synthese nicht erfolgreich abgeschlossen werden kann. Die neue Generation der King Reverse Transkriptase verfügt jedoch über eine einzigartige Strukturdomäne, die die Wasserstoffbrückenbindung zwischen RNA-Strängen zerstören kann, wodurch die komplexe Sekundärstruktur der RNA geöffnet und eine reibungslose reverse Transkription gewährleistet wird.
Alle Produkte können für ODM/OEM angepasst werden. Für Details,Bitte klicken Sie auf kundenspezifischen Service (ODM/OEM)
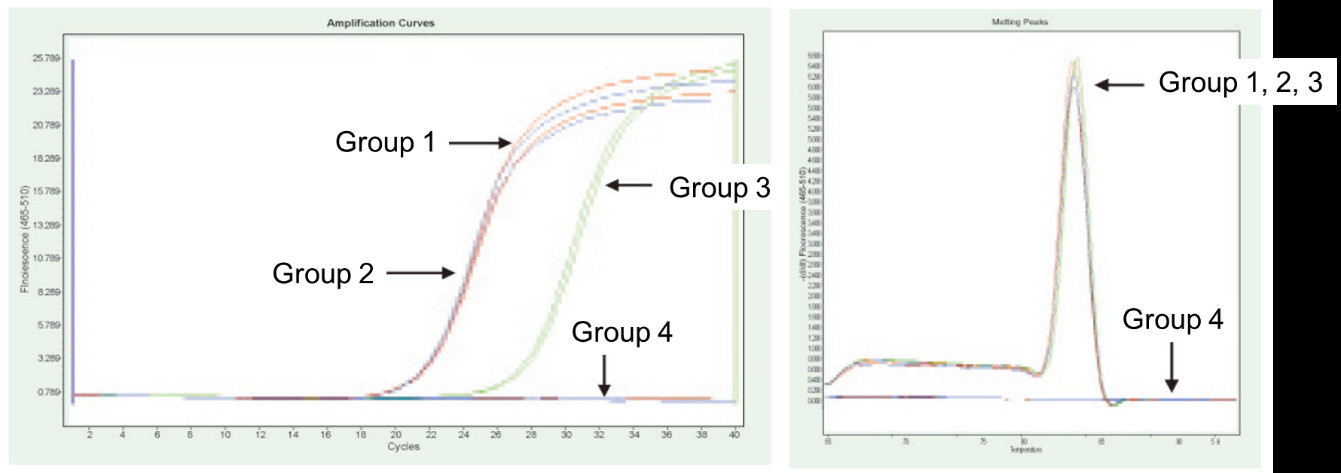 |
Gruppe 1: Reverse Transkription ohne gDNase-Behandlung; Gruppe 2: Keine gDNase-Behandlung und keine reverse Transkription; Gruppe 3: Reverse Transkription nach gDNase-Behandlung; Gruppe 4: gDNase-Behandlung ohne reverse Transkription. Methoden: Quantitativer Fluoreszenz-PCR-Nachweis des TNF-alpha-Gens (Primer entworfen auf Exon mit cDNA oder Genom als Matrize) unter Verwendung von 1 μg Hela-Zell-RNA (mit Genomrest) als Matrize.Ergebnisse: Wie in der Abbildung gezeigt, kann Gruppe 2 reflektieren der Genomrest in RNA, Gruppe 3 kann das wahre Expressionsniveau von TNF-alpha genau widerspiegeln, Gruppe 1 weist Fehler in den endgültigen quantitativen Ergebnissen aufgrund von Genomresten auf und Gruppe 4 zeigt, dass FastKing RT Kit die restliche genomische DNA vollständig entfernen kann RNA. |
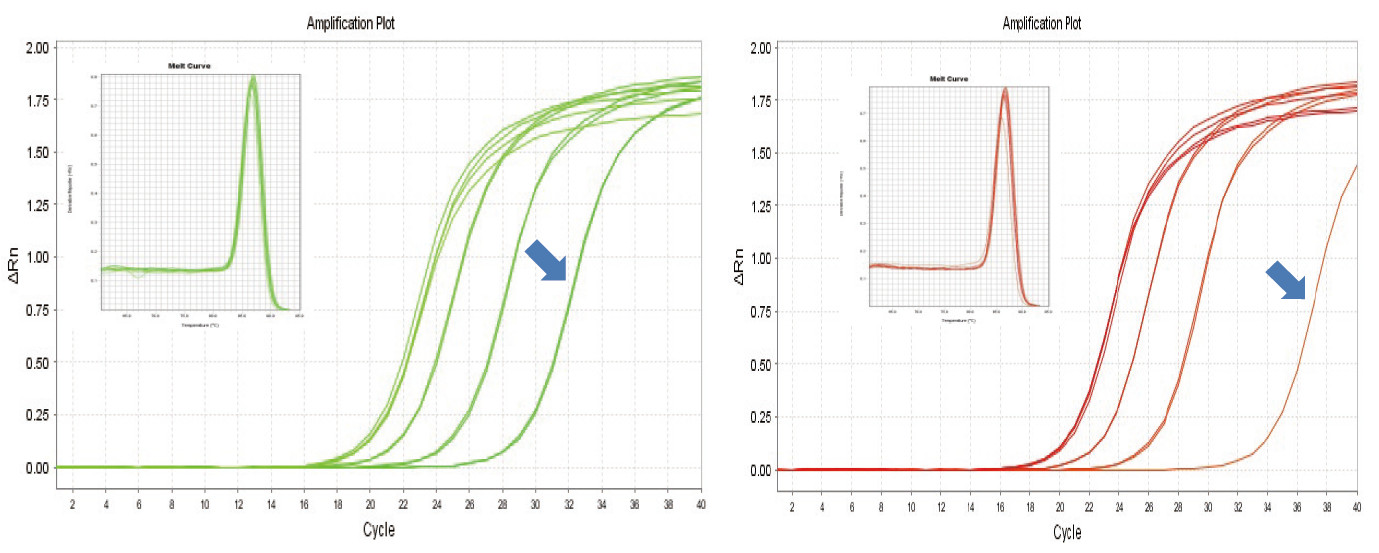 |
Abbildung 1. Die reverse Transkription von Maus-RNA wurde mit dem TIANGEN FastKing RT Kit (links) und dem relevanten Produkt von Lieferant A (rechts) durchgeführt, dann wurde das MM5-Gen quantitativ mit TIANGEN SuperReal PreMix Plus (SYBR Green) amplifiziert. Die Amplifikationskurve und Schmelzkurve wurden analysiert. Der RNA-Input betrug 1000 ng, 100 ng, 10 ng bzw. 1 ng. Die Ergebnisse zeigen, dass das TIANGEN FastKing RT Kit einen klaren reversen Transkriptionsgradienten und einen niedrigen Ct-Wert aufweist und offensichtliche Vorteile für die reverse Transkription von Templates mit geringer Häufigkeit (1 ng, blauer Pfeil) hat. |
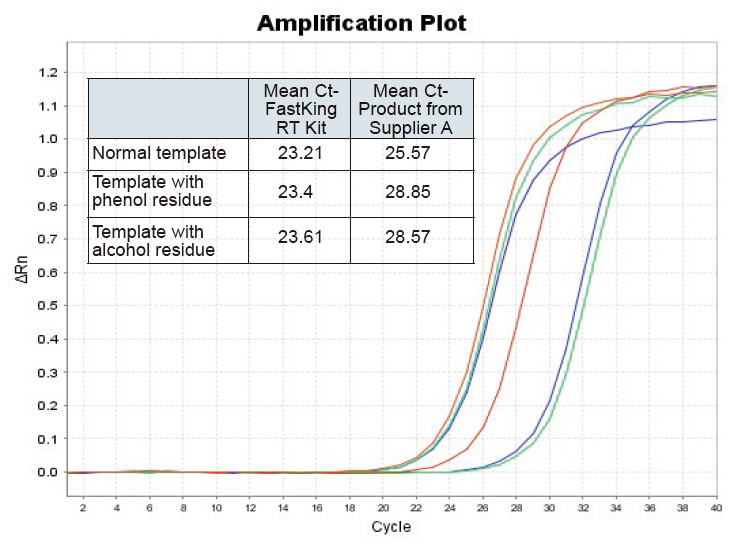 |
Abbildung 2. Reverse Transkription von normaler RNA-Vorlage (rot), Vorlage mit großem Phenolrest (grün) und Vorlage mit Alkoholrest (blau) von Ratten mit dem TIANGEN FastKing RT Kit bzw. dem entsprechenden Produkt von Lieferant A, Quantifizierung der RNC-Gene mit TIANGEN SuperReal PreMix Plus (SYBR Green) und Amplifikationskurven und Ct-Werte wurden analysiert. Die Ergebnisse zeigen, dass das TIANGEN FastKing RT Kit den niedrigsten quantitativen Ct-Wert nach reverser Transkription und eine ausgezeichnete Stressresistenz aufweist und offensichtliche Vorteile für Templates mit hohen Verunreinigungen hat |
A-1-RNA wird abgebaut
——Reinigen Sie hochwertige RNA ohne Kontamination. Das Material, aus dem RNA extrahiert wird, sollte so frisch wie möglich sein, um einen RNA-Abbau zu verhindern. Analysieren Sie die RNA-Integrität auf denaturiertem Gel vor der RT-Reaktion. Nach der RNA-Extraktion sollte es in 100 % Formamid gelagert werden. Wenn ein RNase-Inhibitor verwendet wird, sollte die Erhitzungstemperatur <45°C betragen und der pH-Wert sollte weniger als 8,0 betragen, sonst setzt der Inhibitor die gesamte gebundene RNase frei. Darüber hinaus sollte RNase-Inhibitor in Lösungen mit ≥ 0,8 mM DTT zugesetzt werden.
A-2 RNA enthält Inhibitoren von reversen Transkriptionsreaktionen
——Zu den Inhibitoren der reversen Transkription gehören SDS, EDTA, Glycerin, Natriumpyrophosphat, Spermidin, Formamid, Guanidinsalz usw. Mischen Sie die Kontroll-RNA mit der Probe und vergleichen Sie die Ausbeute mit der Kontroll-RNA-Reaktion, um zu überprüfen, ob ein Inhibitor vorhanden ist. Waschen Sie die RNA-Präzipitation mit 70 % (v/v) Ethanol, um Inhibitoren zu entfernen.
A-3 Unzureichendes Annealing von Primern, die zur Synthese des ersten cDNA-Strangs verwendet werden
——Stellen Sie fest, dass die Annealing-Temperatur für die im Experiment verwendeten Primer geeignet ist. Bei statistischen Hexameren wird empfohlen, die Temperatur 10 min bei 25 °C zu halten, bevor die Reaktionstemperatur erreicht wird. Für genspezifische Primer (GSP), versuchen Sie es mit einem anderen GSP oder wechseln Sie zu Oligo(dT) oder zufälligem Hexamer.
A-4 Kleine Menge Ausgangs-RNA
——Erhöhen Sie die RNA-Menge. Für RNA-Proben unter 50 ng können 0,1 µg bis 0,5 µg Acetyl-BSA in der Erststrang-cDNA-Synthese verwendet werden
A-5 Die Zielsequenz wird in den analysierten Geweben nicht exprimiert.
——Versuchen Sie andere Gewebe.
A-6 PCR-Reaktion schlägt fehl
——Bei der zweistufigen RT-PCR darf die cDNA-Matrize im PCR-Schritt 1/5 des Reaktionsvolumens nicht überschreiten.
A-1 Unspezifisches Annealing von Primern und Templates
——Das 3'-Ende von Primern sollte nicht 2-3 dG oder dC enthalten. Verwenden Sie bei der Erststrangsynthese genspezifische Primer anstelle von Zufallsprimern oder Oligo(dT). Verwenden Sie in den ersten Zyklen eine höhere Glühtemperatur und dann eine niedrigere Glühtemperatur. Verwenden Sie die Hot-Start-Taq-DNA-Polymerase für die PCR, um die Spezifität der Reaktion zu verbessern.
A-2 Schlechtes Design genspezifischer Primer
——Befolgen Sie die gleichen Prinzipien für das Design von Amplifikationsprimern.
A-3 RNA kontaminiert mit genomischer DNA
——Behandeln Sie RNA mit DNase I in PCR-Qualität. Richten Sie eine Kontrollreaktion ohne reverse Transkription ein, um eine DNA-Kontamination zu erkennen.
A-4 Bildung von Primerdimeren
——Entwerfen Sie Primer ohne komplementäre Sequenzen am 3'-Ende.
A-5 Zu hohes Mg2+ Konzentration
——Mg . optimieren2+ Konzentration für jede Template- und Primer-Kombination
A-6 Kontaminiert mit fremder DNA
——Verwenden Sie aerosolresistente Spitzen und UDG-Enzyme.
A-1 Der Gehalt des Erststrangprodukts ist zu hoch
——Reduzieren Sie die Menge des Erststrangprodukts im herkömmlichen PCR-Reaktionsschritt.
A-2 Zu hohe Primermenge in der PCR-Reaktion
——Verringern Sie die Primer-Eingabe.
A-3 Zu viele Zyklen
——Optimieren Sie die PCR-Reaktionsbedingungen und reduzieren Sie die Anzahl der PCR-Zyklen.
A-4 Zu niedrige Glühtemperatur
——Erhöhen Sie die Annealing-Temperatur, um eine unspezifische Initiierung und Verlängerung zu verhindern.
A-5 Unspezifische Amplifikation von Oligonukleotidfragmenten, die durch DNase-Abbau von DNA erzeugt wurden ——Extrahieren Sie hochwertige RNA, um eine DNA-Kontamination zu verhindern.
RT-PCR besteht darin, RNA in cDNA revers zu transkribieren und dann die revers transkribierte cDNA als Matrize für die PCR-Reaktion zu verwenden, um das Zielfragment zu amplifizieren. Wählen Sie entweder Zufallsprimer, Oligo dT und genspezifische Primer gemäß den spezifischen Bedingungen des Experiments. Alle obigen Primer können für kurze eukaryotische Zellen-mRNA ohne Haarnadelstruktur verwendet werden.
Zufallsprimer: Geeignet für lange RNA mit Haarnadelstruktur sowie alle Arten von RNA wie rRNA, mRNA, tRNA usw. Sie werden hauptsächlich für die RT-PCR-Reaktion einzelner Templates verwendet.
Oligo dT: Geeignet für RNA mit PolyA-Tailing (prokaryontische RNA, eukaryotische Oligo dT rRNA und tRNA haben keine PolyA-Tails). Da Oligo dT an den PolyA-Schwanz gebunden ist, muss die Qualität der RNA-Proben hoch sein, und selbst ein geringer Abbau wird die Menge der cDNA-Synthese in voller Länge stark reduzieren.
Genspezifischer Primer: Komplementär zur Matrizensequenz, geeignet für Situationen, in denen die Zielsequenz bekannt ist.
Es gibt zwei Möglichkeiten:
1. Interne Referenzmethode: Theoretisch handelt es sich bei cDNA um DNA-Fragmente unterschiedlicher Länge, daher ist das Ergebnis der Elektrophorese ein Abstrich. Wenn die RNA-Häufigkeit gering ist, wird in der Elektrophorese kein Produkt angezeigt, dies bedeutet jedoch nicht, dass kein Produkt durch PCR amplifiziert wird. Im Allgemeinen kann eine interne Referenz verwendet werden, um cDNA nachzuweisen. Wenn die interne Referenz Ergebnisse liefert, kann die Qualität der cDNA grundsätzlich gewährleistet werden (in wenigen Fällen kann es bei zu langen Zielgenfragmenten Ausnahmen geben).
2. Wenn ein bekanntes Gen durch diese Matrize amplifiziert wird, kann es durch die Primer dieses Gens verifiziert werden. Die Amplifikation der internen Referenz bedeutet nicht unbedingt, dass es kein Problem mit cDNA gibt. Da die interne Referenz eine hohe Häufigkeit an cDNA aufweist, ist sie leicht zu amplifizieren. Wenn cDNA aus verschiedenen Gründen teilweise abgebaut wird, werden aus Sicht der Wahrscheinlichkeit PCR-Ergebnisse von Zielgenen mit geringer Häufigkeit stark beeinflusst. Während die interne Referenz immer noch im Überfluss vorhanden ist, wird die Verstärkung wahrscheinlich nicht beeinträchtigt.
Teilweise Abbau von RNA. Erkennen Sie die Integrität und reinigen Sie die RNA
Die RNA-Gehalte verschiedener Spezies können unterschiedlich sein, aber im Allgemeinen sollte die extrahierte Gesamt-RNA in der Gelelektrophorese zwei klare 28S- und 18S-Banden enthalten, und die Helligkeit der ersteren Bande sollte doppelt so hoch sein wie die der letzteren. Die 5S-Bande zeigt an, dass RNA abgebaut wurde, und ihre Helligkeit ist proportional zum Grad des Abbaus. Die erfolgreiche Amplifikation der internen Referenz bedeutet nicht, dass es kein Problem mit RNA gibt, da die interne Referenz in großer Menge vorhanden ist und RNA amplifiziert werden kann, solange der Abbau nicht schwerwiegend ist. Die OD260/OD280Der Anteil an reiner RNA, gemessen mit einem Spektrophotometer, sollte zwischen 1,9 und 2,1 liegen. Eine geringe Menge an Proteinverunreinigung in der RNA verringert das Verhältnis. Solange der Wert nicht zu niedrig ist, wird RT nicht beeinflusst. Was für RT am wichtigsten ist, ist die RNA-Integrität.
Die Verlängerung des internen Referenzgens kann nur anzeigen, dass die RT erfolgreich war, hängt aber nicht unbedingt mit der Qualität des cDNA-Strangs zusammen. Da die internen Referenzfragmente im Allgemeinen eine kleine Größe und eine hohe Expression aufweisen, sind sie bei der reversen Transkription leichter erfolgreich. Die Größe und Expression des Zielgens variiert jedoch von Gen zu Gen. Die cDNA-Qualität kann nicht nur durch interne Referenz beurteilt werden, insbesondere für die Zielfragmente, die länger als 2 kb sind.
Einige Proben haben komplexe Sekundärstrukturen oder einen hohen GC-Gehalt oder sind kostbar mit geringer Häufigkeit. In diesen Fällen sollte die geeignete reverse Transkriptase entsprechend der Größe des Zielfragments und der Probe ausgewählt werden. Bei RNA-Matrizen mit hohem GC-Gehalt und komplexer Sekundärstruktur ist es schwierig, die Sekundärstruktur bei niedriger Temperatur oder mit üblicher reverser Transkriptase zu öffnen. Für diese Matrizen kann Quant Reverse Transcriptase ausgewählt werden, da ihre reverse Transkriptionsleistung offensichtlich besser ist als die der reversen Transkriptase der M-MLV-Serie, die verschiedene RNA-Matrizen effizient revers transkribieren und RNA maximal in cDNA-Erststrang transkribieren kann. Bei Verwendung des allgemeinen Reverse-Transkriptase-Kits kann das 20-μl-System nur 1 μg Gesamt-RNA effektiv revers transkribieren. Bitte beachten Sie die maximale RT-Kapazität des Kits. Wenn die Matrize im Überschuss hinzugefügt wird, begünstigt die reverse Transkription die RNA mit hoher Häufigkeit. Daher ist es besser, die maximale Kapazität des Systems nicht zu überschreiten.
A-1 Stellen Sie fest, ob die RNA stark abgebaut ist und ob die RT erfolgreich ist
Im Allgemeinen wird der Grund für das Versagen der internen Referenzamplifikation oft durch einen ernsthaften RNA-Abbau verursacht. Ein weiterer möglicher Grund ist das Versagen der reversen Transkription. Die interne Referenz kann nicht als Standard zur Beurteilung der Qualität eines cDNA-Einzelstrangs verwendet werden, kann jedoch als Standard verwendet werden, um zu beurteilen, ob die reverse Transkription erfolgreich ist, wenn kein Problem mit der RNA-Qualität vorliegt. Das Wichtigste beim reversen Transkriptionsprozess ist die Aufrechterhaltung einer konstanten Temperatur und eines konstanten Reaktionssystems, um die Reaktionseffizienz zu verbessern.
A-2 Bestimmen Sie, ob die Primer zur Amplifikation interner Referenzgene zuverlässig sind und ob es Probleme mit den in der PCR verwendeten Reagenzien gibt.
Zur relativen Quantifizierung muss die RNA vor der reversen Transkription quantifiziert werden, was auch in vielen Kits für die reverse Transkription erforderlich ist, z. B. quantifizieren Sie den RNA-Input als 1 μg. Da die revers transkribierte cDNA eine gemischte Lösung ist, die RNA, Oligo-dT, Enzym, dNTP und sogar einen kleinen DNA-Rest enthält, wird eine Abweichung verursacht, so dass es unmöglich ist, die cDNA genau zu quantifizieren. Daher ist eine RNA-Quantifizierung erforderlich. Wenn man bedenkt, dass die Effizienz der reversen Transkription bei verschiedenen Proben gleich ist, sollte die erhaltene cDNA-Menge gleich sein, und die quantitative Analyse kann den Vergleich der Expressionsniveaus verschiedener Gene in derselben Menge an Gesamt-RNA zeigen. Bei der Durchführung einer quantitativen relativen Fluoreszenz-PCR ist nach der reversen Transkription möglicherweise keine quantitative cDNA erforderlich, da das interne Referenzgen als Referenz dienen kann.
Es hängt hauptsächlich mit den Genen zusammen, und die reverse Transkription langer Fragmente ist für die meisten Gene nicht möglich. Erstens ist die Effizienz der reversen Transkription viel geringer als die der PCR. Zweitens schränken die GC-reiche Region und die Sekundärstruktur vieler Gene sowohl die reverse Transkription als auch die PCR ein. Schließlich sind die Genauigkeit und die Amplifikationseffizienz der PCR gleichzeitig schwer zu garantieren. Bei der reversen Transkription kann niemand garantieren, lange Fragmente für Gene mit geringer Kopienzahl zu erhalten, insbesondere unter Verwendung von Oligo-dT. Bei 5' UTR mit mehr GC ist es noch schwieriger. Daher ist es immer noch eine vernünftige Methode, das Transkript mit zufälligen Primern zu reversieren, die natürlichen Schnittstellen im Zielfragment zu finden, segmentweise zu amplifizieren und dann den Restriktionsverdau und die Ligation durchzuführen. Im Allgemeinen ist es schwierig, Fragmente, die größer als 2 kb sind, direkt zu amplifizieren, aber es ist nicht immer unmöglich, Folgendes zu erhalten: 1. Zuallererst die Integrität der RNA/mRNA garantieren, und die TRIZOL-Extraktion wird bevorzugt. 2.M-MLV RT-PCR-Kit kann direkt verwendet werden. Verlängern Sie die Annealing-Zeit und erhöhen Sie die Zykluszahl im Amplifikationsprozess richtig. Alternativ kann eine verschachtelte PCR angewendet werden oder zuerst eine oder zwei Reaktionen mit entsprechend verlängerter Denaturierungs- und Verlängerungszeit vor der normalen PCR-Amplifikation durchgeführt werden, was zur Verlängerung von Fragmenten beitragen kann. Achten Sie auf die Treue der Polymerase. 3.Long Taq kann in der PCR verwendet werden, um ideale Ergebnisse zu erzielen. 4.Für die Anwendung der Proteinexpression sollte High-Fidelity-Polymerase verwendet werden.
TIANGEN bietet zwei Arten von reverser Transkriptase an: Quant/King RTase und TIANScript M-MLV. Der Hauptunterschied zwischen ihnen ist die Eingabemenge der Vorlagen. Quant ist eine einzigartige reverse Transkriptase, die sich von dem üblicherweise verwendeten M-MLV unterscheidet, das aus dem Moloney-Maus-Leukämievirus stammt. Quant ist eine neue hocheffiziente Reverse Transkriptase, die rekombinant durch manipulierte Escherichia coli exprimiert wird. Quant eignet sich zur Amplifikation von 50 ng-2 µg RNA mit hoher reverser Transkriptionsaktivität und hoher Ausbeute. Im Vergleich zu gewöhnlichem MMLV oder AMV besteht die größte Eigenschaft von Quant darin, dass es eine sehr starke Affinität zu RNA-Matrizen aufweist und Transkript-Komplex-Matrizen ohne Denaturierung bei hoher Temperatur umkehren kann. Bei Templaten mit höherem GC-Gehalt ist die umgekehrte Effizienz höher. Diese reverse Transkriptase weist jedoch RNase H-Aktivität auf, die die Länge des cDNA-Produkts beeinflussen kann (geeignet für < 4,5 kb-Matrizen). Für die konventionelle Reverse Transkription wird die TIANScript MMLV Reverse Transkriptase empfohlen. Diese RTase ist ein modifiziertes Enzym mit sehr schwacher RNase H-Aktivität, das für lange (> 5 kb) cDNA-Synthese geeignet ist.
Die Reverse Transkription und die PCR-Amplifikation in einem Schritt werden im selben Röhrchen durchgeführt, ohne dass der Röhrchendeckel zwischen cDNA-Synthese und Amplifikation geöffnet werden muss, was zur Reduzierung der Kontamination hilfreich ist. Da alle erhaltenen cDNA-Proben zur Amplifikation verwendet werden, ist die Sensitivität mit einem Minimum von 0,01 pg Gesamt-RNA höher. Für eine erfolgreiche einstufige RTPCR werden im Allgemeinen genspezifische Primer verwendet, um die cDNA-Synthese zu initiieren. Das zweistufige Verfahren, nämlich reverse Transkription und PCR-Amplifikation, wird in zwei Schritten durchgeführt. Zunächst wird eine reverse Transkription von einer RNA-Matrize durchgeführt, um cDNA zu erhalten, und die erhaltene cDNA wird einer oder mehreren unterschiedlichen PCR-Reaktionen unterzogen. Das zweistufige Verfahren kann Oligo(dT)- oder Zufallsprimer verwenden, um die Synthese des ersten cDNA-Strangs zu steuern, und kann alle mRNA-Informationen aus einer spezifischen Probe revers transkribieren.
Produktkategorien
WARUM UNS WÄHLEN
Seit seiner Gründung entwickelt unsere Fabrik erstklassige Produkte unter Einhaltung des Prinzips
der Qualität zuerst. Unsere Produkte haben einen ausgezeichneten Ruf in der Branche und ein wertvolles Vertrauen bei neuen und alten Kunden gewonnen.










