2×Taq Platinum PCR-Mix
Aktivitätsdefinition
1 Einheit (E) Taq Platinum DNA Polymerase-Aktivität ist definiert als die Enzymmenge, die erforderlich ist, um 10 nmol Desoxynukleotide in säureunlösliche Substanzen bei 74 °C innerhalb von 30 min unter Verwendung von aktivierter Lachssperma-DNA als Matrize/Primer einzubauen.
Qualitätskontrolle
Die Reinheit durch SDS-PAGE-Nachweis beträgt mehr als 99%; Es wird keine Aktivität der exogenen Nuklease nachgewiesen; Single-Copy-Gen im menschlichen Genom könnte effektiv amplifiziert werden; Keine signifikante Aktivitätsänderung bei Lagerung bei Raumtemperatur für eine Woche.
Haupttechnische Parameter
Es hat 5'-3'-Exonuklease-Aktivität und 3'-5'-Exonuklease-Aktivität, und seine Treue ist der Pfu-Polymerase nahe. Die Verlängerungsgeschwindigkeit von Taq Platinum Polymerase ist schneller als die von Pfu Polymerase und die Amplifikationseffizienz ist höher. PCR-Produkte können direkt an das glatte Ende ligiert oder mit dem TA-Vektor kloniert werden. Wenn die Klonierungseffizienz verbessert werden muss, wird empfohlen, zuerst zu reinigen und 3'-dA-Überhänge hinzuzufügen, bevor in den TA-Vektor kloniert wird.
Taq Platinum MasterMix mit einer Röhre (Nationale High-Tech-Produktzertifizierung)
■ Der Taq Platinum MasterMix hat eine verbesserte Spezifität und Sensitivität der PCR-Reaktion und kann komplexe Template mit hohem GC-Gehalt, Sekundärstruktur und dergleichen amplifizieren. Es können nur 2 Kopien des Zieltemplates amplifiziert werden, was genauere experimentelle Ergebnisse gewährleistet.
■ Die einzigartige Taq Platinum MasterMix-Formel macht das gesamte Reaktionssystem sehr stabil und die Aktivität wird nicht durch wiederholtes Einfrieren/Auftauen oder Langzeitlagerung bei 4 °C beeinträchtigt.
■ Die stabile und effiziente vorgefertigte PCR-Mischlösung kann die Operation schnell und einfach machen, wodurch Arbeitsintensität und Probenahmefehler stark reduziert werden. Ebenfalls im Mix enthalten sind Hochleistungs-PCR-Enhancer und -Optimierer, was die Anforderungen an die PCR-Bedingungen reduziert.
■ Dieses Produkt enthält sowohl farbstoffhaltige als auch farbstofffreie Systeme. Farbstoffhaltige MasterMix-Produkte können nach der PCR direkt elektrophoresiert werden, ohne dass Ladepuffer hinzugefügt werden muss.
Anwendungen
Es kann die Pfu-Polymerase ersetzen, um High-Fidelity-Produkte aus komplexen Templaten wie Genomen zu amplifizieren, und es eignet sich für Anwendungen wie das Klonen von Expressionsgenen, ortsspezifische Mutationen und die Analyse von Einzelnukleotidpolymorphismus (SNP) usw.
Vorsichtsmaßnahmen beim Entwerfen von PCR-Primern:
Die Primerlänge beträgt normalerweise 20-25 mer. Wenn jedoch eine PCR mit langen Fragmenten durchgeführt wird, sollte die Primerlänge auf 30-35-mer erhöht werden.
■ Es gibt keine komplementäre Paarung zwischen den beiden Primern, insbesondere für die letzten 3 Basen am 3'-Ende.
■ Der GC-Gehalt sollte 50-60 % betragen und lokale reiche GC oder AT vermeiden. Um Primer und Template stabil zu binden, vermeiden Sie eine AT-reiche Struktur am 3'-Ende.
■ Grundierung zur Bildung einer Sekundärstruktur vermeiden.
■ Wählen Sie zwei Primer mit nahe beieinander liegenden Tm-Temperaturen.
Berechnung des Tm-Wertes von Primern für die PCR:
■ Wenn der Primer weniger als 20 mer beträgt: Tm = 2 °C × (A+T) + 4 °C × (G+C).
■ Wenn der Primer mehr als 20 mer hat: Tm = 81,5 + 0,41 × (GC%) – 600/l, wobei L die Länge des Primers ist.
■ Stellen Sie die Glühtemperatur auf (Tm-5)°C ein.
PCR-Primer-Eingabe
Die geeignete Endkonzentration der Primer kann zwischen 0,1 µM und 1,0 µM gewählt werden. Eine zu niedrige Primerkonzentration führt zu einer geringen Ausbeute an Amplifikationsprodukten, während eine zu hohe Primerkonzentration anfälliger für unspezifische Amplifikation ist. Wenn die Menge an Matrizen-DNA groß ist oder komplexe Matrizen-DNA (wie menschliche Genom-DNA) als Matrize verwendet wird, sollte die Primerkonzentration normalerweise niedriger sein. Wenn die Menge an Matrizen-DNA klein ist oder einfache Matrizen-DNA (zB Plasmid-DNA usw.) als Matrize verwendet wird, sollte die Primerkonzentration höher sein.
Alle Produkte können für ODM/OEM angepasst werden. Für Details,Bitte klicken Sie auf kundenspezifischen Service (ODM/OEM)
 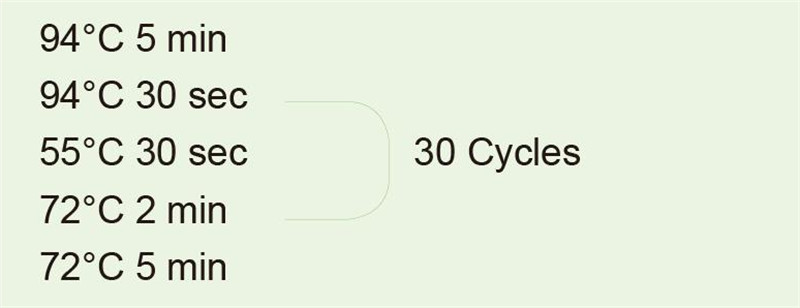 |
Verwenden Sie genomische DNA als Matrize, um das 1-kb-Fragment zu amplifizieren. Nehmen Sie nach der PCR-Reaktion 5 μl für den Elektrophorese-Nachweis. |
A-1 Vorlage
■ Das Template enthält Proteinverunreinigungen oder Taq-Inhibitoren usw. ——Reinigen Sie das DNA-Template, entfernen Sie Proteinverunreinigungen oder extrahieren Sie Template-DNA mit Reinigungskits.
■ Die Denaturierung des Templates ist nicht abgeschlossen ——Erhöhen Sie die Denaturierungstemperatur entsprechend und verlängern Sie die Denaturierungszeit.
■ Vorlagenverschlechterung – – Bereiten Sie die Vorlage erneut vor.
A-2 Grundierung
■ Schlechte Qualität der Grundierungen ——Resynthetisieren Sie die Grundierung.
■ Primerabbau ——Aliquotieren Sie die hochkonzentrierten Primer zur Konservierung in ein kleines Volumen. Mehrfaches Einfrieren und Auftauen oder Langzeit-Kryokonservierung bei 4 °C vermeiden.
■ Unsachgemäßes Design von Primern (zB Primerlänge nicht ausreichend, Dimer zwischen Primern gebildet usw.) -Redesign von Primern (Vermeidung der Bildung von Primer-Dimer und Sekundärstruktur)
A-3 mg2+Konzentration
■ Mg2+ Konzentration ist zu niedrig ——Mg . richtig erhöhen2+ Konzentration: Optimierung des Mg2+ Konzentration durch eine Reihe von Reaktionen von 1 mM bis 3 mM mit einem Intervall von 0,5 mM, um das optimale Mg . zu bestimmen2+ Konzentration für jede Matrize und jeden Primer.
A-4 Glühtemperatur
■ Die hohe Annealing-Temperatur beeinflusst die Bindung von Primer und Template. ——Reduzieren Sie die Glühtemperatur und optimieren Sie den Zustand mit einem Gradienten von 2°C.
A-5 Verlängerungszeit
■ Kurze Verlängerungszeit——Verlängert die Verlängerungszeit.
Phänomene: Negative Proben zeigen auch die Zielsequenzbanden.
A-1 Kontamination von PCR
■ Kreuzkontamination von Zielsequenz- oder Amplifikationsprodukten ——Die Probe, die die Zielsequenz enthält, vorsichtig in die negative Probe pipettieren oder aus dem Zentrifugenröhrchen verschütten. Die Reagenzien oder Geräte sollten autoklaviert werden, um vorhandene Nukleinsäuren zu eliminieren, und das Vorliegen einer Kontamination sollte durch Negativkontrollexperimente bestimmt werden.
■ Kontamination mit Reagenzien ——Aliquotieren Sie die Reagenzien und lagern Sie sie bei niedriger Temperatur.
A-2 Primer
■ Mg2+ Konzentration ist zu niedrig ——Mg . richtig erhöhen2+ Konzentration: Optimierung des Mg2+ Konzentration durch eine Reihe von Reaktionen von 1 mM bis 3 mM mit einem Intervall von 0,5 mM, um das optimale Mg . zu bestimmen2+ Konzentration für jede Matrize und jeden Primer.
■ Unsachgemäßes Primer-Design und die Zielsequenz weist Homologie mit der Nicht-Zielsequenz auf. ——Grundierungen neu gestalten.
Phänomene: Die PCR-Amplifikationsbanden stimmen nicht mit der erwarteten Größe überein, entweder groß oder klein, oder manchmal treten sowohl spezifische Amplifikationsbanden als auch unspezifische Amplifikationsbanden auf.
A-1 Grundierung
■ Schlechte Primer-Spezifität
——Grundierung neu gestalten.
■ Die Primerkonzentration ist zu hoch ——Erhöhen Sie die Denaturierungstemperatur richtig und verlängern Sie die Denaturierungszeit.
A-2 mg2+ Konzentration
■ Das Mg2+ Konzentration zu hoch ——Mg2+ Konzentration richtig reduzieren: Mg . optimieren2+ Konzentration durch eine Reihe von Reaktionen von 1 mM bis 3 mM mit einem Intervall von 0,5 mM, um das optimale Mg . zu bestimmen2+ Konzentration für jede Matrize und jeden Primer.
A-3 Thermostabile Polymerase
■ Übermäßige Enzymmenge ——Verringern Sie die Enzymmenge entsprechend in Intervallen von 0,5 U.
A-4 Glühtemperatur
■ Die Glühtemperatur ist zu niedrig ——Erhöhen Sie die Glühtemperatur entsprechend oder wenden Sie die zweistufige Glühmethode an
A-5 PCR-Zyklen
■ Zu viele PCR-Zyklen ——Reduzieren Sie die Anzahl der PCR-Zyklen.
A-1 Grundierung——Schlechte Spezifität ——Entwerfen Sie den Primer neu, ändern Sie die Position und Länge des Primers, um seine Spezifität zu verbessern; oder verschachtelte PCR durchführen.
A-2 Template-DNA
——Die Matrize ist nicht rein ——Reinigen Sie die Matrize oder extrahieren Sie DNA mit Aufreinigungskits.
A-3 mg2+ Konzentration
——Mg2+ Konzentration zu hoch ——Mg . richtig reduzieren2+ Konzentration: Optimierung des Mg2+ Konzentration durch eine Reihe von Reaktionen von 1 mM bis 3 mM mit einem Intervall von 0,5 mM, um das optimale Mg . zu bestimmen2+ Konzentration für jede Matrize und jeden Primer.
A-4 dNTP
——Die dNTP-Konzentration ist zu hoch ——Reduzieren Sie die dNTP-Konzentration entsprechend
A-5 Glühtemperatur
——Zu niedrige Glühtemperatur ——Glühtemperatur entsprechend erhöhen
A-6 Zyklen
——Zu viele Zyklen ——Optimieren Sie die Zyklenzahl

Der erste Schritt besteht darin, die geeignete Polymerase auszuwählen. Reguläre Taq-Polymerase kann aufgrund fehlender 3'-5'-Exonuklease-Aktivität nicht Korrektur gelesen werden, und eine Fehlpaarung verringert die Verlängerungseffizienz von Fragmenten stark. Daher kann normale Taq-Polymerase Zielfragmente, die größer als 5 kb sind, nicht effektiv amplifizieren. Taq-Polymerase mit spezieller Modifikation oder eine andere High-Fidelity-Polymerase sollte ausgewählt werden, um die Verlängerungseffizienz zu verbessern und die Anforderungen der Amplifikation langer Fragmente zu erfüllen. Außerdem erfordert die Amplifikation langer Fragmente auch eine entsprechende Anpassung von Primerdesign, Denaturierungszeit, Extensionszeit, Puffer-pH usw. Üblicherweise können Primer mit 18-24 bp zu einer besseren Ausbeute führen. Um Matrizenschäden zu vermeiden, sollte die Denaturierungszeit bei 94 °C auf 30 Sekunden oder weniger pro Zyklus reduziert werden und die Zeit zum Temperaturanstieg auf 94 °C vor der Amplifikation sollte weniger als 1 Minute betragen. Darüber hinaus kann eine effektive Amplifikation langer Fragmente durch Einstellen der Extensionstemperatur auf etwa 68 °C und Gestaltung der Extensionszeit entsprechend der Rate von 1 kb/min sichergestellt werden.
Die Fehlerrate der PCR-Amplifikation kann durch die Verwendung verschiedener DNA-Polymerasen mit hoher Genauigkeit reduziert werden. Unter allen bisher gefundenen Taq-DNA-Polymerasen hat das Pfu-Enzym die niedrigste Fehlerrate und die höchste Genauigkeit (siehe beigefügte Tabelle). Neben der Enzymauswahl können Forscher die PCR-Mutationsrate weiter reduzieren, indem sie die Reaktionsbedingungen optimieren, einschließlich der Optimierung der Pufferzusammensetzung, der Konzentration der thermostabilen Polymerase und der Optimierung der PCR-Zykluszahl.
Produktkategorien
WARUM UNS WÄHLEN
Seit seiner Gründung entwickelt unsere Fabrik erstklassige Produkte unter Einhaltung des Prinzips
der Qualität zuerst. Unsere Produkte haben einen ausgezeichneten Ruf in der Branche und ein wertvolles Vertrauen bei neuen und alten Kunden gewonnen.








